
В первом семестре я работал с поли(А)-полимеразой Aquifex aerocus. Данный белок содержит 824 аминокислотных остатков и кодируется 2474 нуклеотидами.
Для этого откроем сервис CDD и вставим последовательность белка в FASTA-формате в поле поиска. Необходимо выбрать режим Full Results.
Таким образом я получил большой список хитов(реально большой, нет смысла приводить его здесь). Из них я отобрал 10 хитов, относящих мой белок к тому или КОГу.
| ID | E-value | Координаты | Название на английском | Название на русском | Функциональная категория на английском | Функциональная категория на русском |
| PcnB(COG0617) | 7.41e-73 | 430-823 | tRNA nucleotidyltransferase/poly(A) polymerase | ТРНК нуклеотидилтрансфераза/поли(А) полимераза | Translation, ribosomal structure and biogenesis | Транскрипция, структура рибосом и биогенез |
| NrnA(COG0618) | 1.40e-57 | 1-299 | nanoRNase/pAp phosphatase, hydrolyzes c-di-AMP and oligoRNAs | наноРНКаза/pAp-фосфатаза, гидролаза c-ди-AMФ и олигонуклеотидов | Nucleotide transport and metabolism | Транспорт нуклеотидов и метаболизм |
| GdpP(COG3887) | 7.22e-16 | 14-295 | c-di-AMP phosphodiesterase, consists of a GGDEF-like and DHH domains | с-ди-АМФ фосфодиэстераза, состоящая из доменов GGDEF и DHH | Signal transduction mechanisms | Механизмы передачи сигналов |
| CBS(COG0517) | 1.00e-14 | 301-413 | CBS domain | CBS-домен | Signal transduction mechanisms | Механизмы передачи сигналов |
| COG3620 | 3.84e-10 | 284-418 | Predicted transcriptional regulator with C-terminal CBS domains | Прогнозируемый регулятор транскрипции, содержащий C-терминальные CBS-домены | Transcription | Транскрипция |
| COG2905 | 7.83e-10 | 273-450 | Signal-transduction protein containing cAMP-binding, CBS, and nucleotidyltransferase domains | Сигнально-трансдукционный белок, содержащий cAMФ-связывающие, CBS и нуклеотидилтрансферазные домены | Signal transduction mechanisms | Механизмы передачи сигналов |
| PPX1(COG1227) | 2.04e-08 | 11-176 | Inorganic pyrophosphatase/exopolyphosphatase | Пирофосфатаза/экзополифосфатаза | Energy production and conversion, Inorganic ion transport and metabolism | Производство и преобразование энергии, транспорт нерганических ионов и метаболизм |
| YtoI(COG4109) | 8.62e-08 | 281-439 | Predicted transcriptional regulator containing CBS domains | Прогнозируемый регулятор транскрипции, содержащий CBS-домены | Transcription | Транскрипция |
| MgtE(COG2239) | 1.33e-07 | 304-426 | Mg/Co/Ni transporter MgtE (contains CBS domain) | Mg/Co/Ni транспортер MgtE, содержащий домен CBS | Inorganic ion transport and metabolism | Tранспорт нерганических ионов и метаболизм |
| COG3448 | 2.39e-07 | 292-420 | CBS-domain-containing membrane protein | Мембранный белок, содержащий CBS-домен | Signal transduction mechanisms | Механизмы передачи сигналов |
| COG2524 | 8.56e-07 | 298-416 | Predicted transcriptional regulator, contains C-terminal CBS domains | Прогнозируемый регулятор транскрипции, содержащий C-терминальные CBS-домены | Transcription | Транскрипция |
| ProV(COG4175) | 1.02e-03 | 299-416 | ABC-type proline/glycine betaine transport system, ATPase component | АТФазный компонент пролин/глициновой бетаин-транспортной системы АВС-типа | Amino acid transport and metabolism | Транспорт аминокислот и метаболизм |
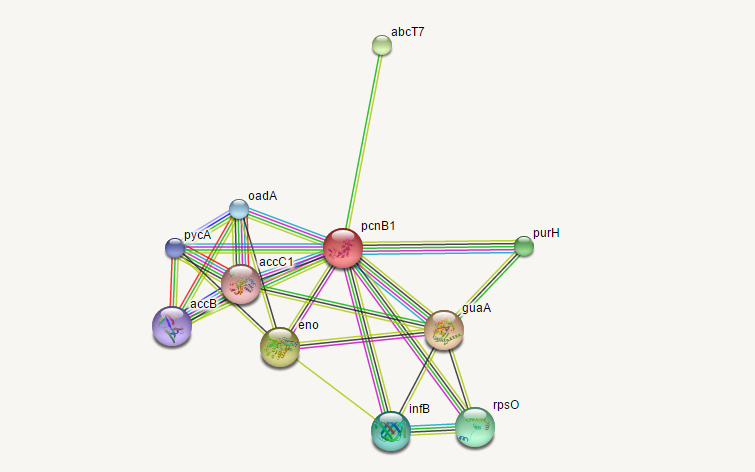
Получим изображение геномного окружения для КОГа c наилучшим e-value с помощью сервиса STRING
Я выбрал КОГ COG0617. Результаты по нему приведены ниже. Параметры программы были взяты по умолчанию: максимальное количество взаимодействующих по первой линии белков (first shell interactors) - 10, минимальный требуемый score взаимодействия - 0.400.


Каждый узел графа символизирует совокупность всех изоформ белка, транскрибирующихся с одного и того же локуса гена. Ребра графа символизируют белок-белковые взаимосвязи (физическое соединение или даже слияние, коэкспрессия, совместная функция). Белки, для которых известна 3D-структура отображены большимим узлами с соответсвующим изображением внутри, остальные белки отображены узлами меньшего размера.
Видно, что некоторая часть взаимосвязей не является достоверной (ребра зеленого цвета), однако большая часть взаимосвязей экспериментально доказана (фиолетовое ребро, 9 штук).
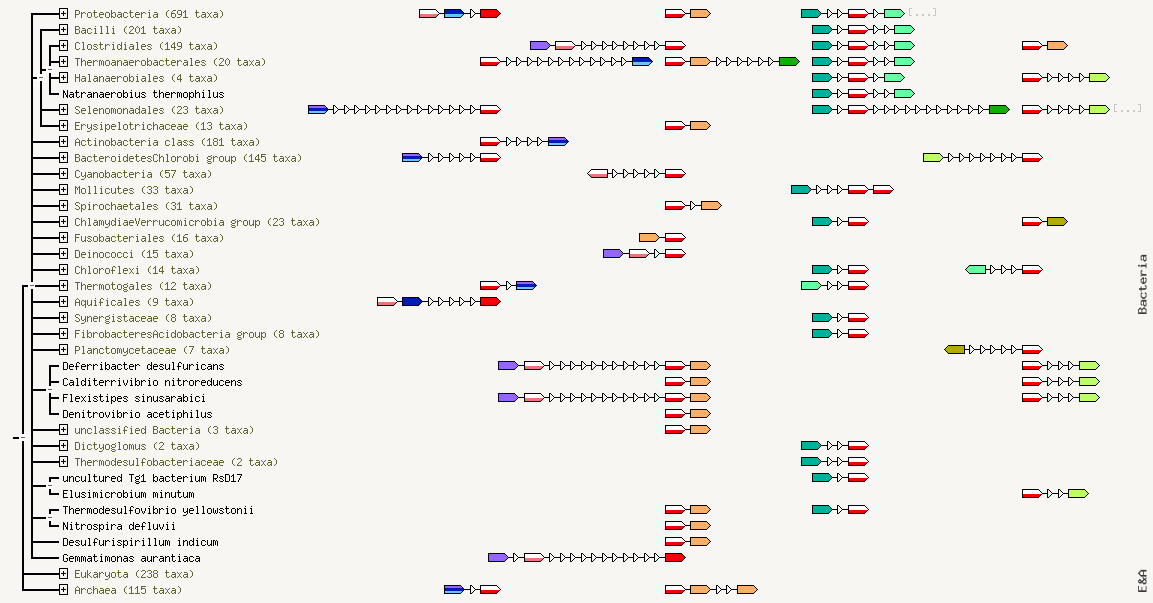


Полученное изображение очень велико, я не вижу смысла приводить здесь его целиком.
Видно, что эти белки встречаются вместе (за редким исключением) у многих бактерий.
С помощью инструмента AmiGO (раздел Tools/Resourses, Analysis of GO data, BLAST) я обнаружил белок, который наиболее похож на исходный. Для этого я ввел в окно поиска последовательность моей поли(А)-полимеразы. Лучшая находка - поли(А)-полимераза бактерии Geobacter sulfurreducens PCA, e-value = 8.6e-103, что является довольно хорошим результатом.
Идентификатор найденного белка GSU_1581, я считаю что термины GO можно переносить с него на мой белок

Выравнивание показывает, что эти белки имеют много гомологичных участков.
| Аспект | Идентификатор GO | Название термина | Перевод названия термина | Код типа достоверности |
| Биологический процесс (biological process) | GO:0006396 | RNA processing | Процессинг РНК | ISS |
| Биологическая функция (molecular function) | GO:0004652 | Polynucleotide adenylyltransferase activity | Активность полинуклеотид аденилилтрансферазы | ISS |
| Биологическая функция (molecular function) | GO:0003723 | RNA binding | Связывание РНК | ISS |
© Борисов Евгений 2017